Abstract
智能医学图像分割方法正在快速地发展和应用,但面临着域转移挑战,即由于源域和目标域数据分布不同导致算法性能下降。为此,本文提出了一种基于生成对抗网络(GAN)的无监督端到端域自适应医学图像分割方法。设计网络训练调整模型,由分割网络和鉴别网络组成。分割网络以残差模块为基本模块,增加对特征的复用能力,降低模型优化难度,并将分割损失与对抗损失相结合,在鉴别网络的作用下学习图像特征层面的跨域特征。鉴别网络采用卷积神经网络,并带入源域标签训练,用来区分生成网络的分割结果是来自源域或目标域,整个训练过程无监督。使用膝关节磁共振(MR)图像公开数据集和采集的临床数据集进行实验,与经典的特征级域自适应方法和图像级域自适应方法对比,所提方法的平均戴斯相似性系数(DSC)分别提高了2.52%与6.10%。本文方法有效提高了分割方法的域自适应能力,显著提高了对胫骨和股骨的分割精度,可以较好地解决磁共振图像分割中的域转移问题。
Keywords: 医学图像分割, 域转移, 生成对抗网络, 图像级域自适应, 特征级域自适应
Abstract
Intelligent medical image segmentation methods have been rapidly developed and applied, while a significant challenge is domain shift. That is, the segmentation performance degrades due to distribution differences between the source domain and the target domain. This paper proposed an unsupervised end-to-end domain adaptation medical image segmentation method based on the generative adversarial network (GAN). A network training and adjustment model was designed, including segmentation and discriminant networks. In the segmentation network, the residual module was used as the basic module to increase feature reusability and reduce model optimization difficulty. Further, it learned cross-domain features at the image feature level with the help of the discriminant network and a combination of segmentation loss with adversarial loss. The discriminant network took the convolutional neural network and used the labels from the source domain, to distinguish whether the segmentation result of the generated network is from the source domain or the target domain. The whole training process was unsupervised. The proposed method was tested with experiments on a public dataset of knee magnetic resonance (MR) images and the clinical dataset from our cooperative hospital. With our method, the mean Dice similarity coefficient (DSC) of segmentation results increased by 2.52% and 6.10% to the classical feature level and image level domain adaptive method. The proposed method effectively improves the domain adaptive ability of the segmentation method, significantly improves the segmentation accuracy of the tibia and femur, and can better solve the domain transfer problem in MR image segmentation.
Keywords: Medical image segmentation, Domain shift, Generative adversarial networks, Image level domain adaptation, Feature level domain adaptation
引言
医学图像分割是决定医学图像在临床诊疗中能否提供可靠依据的关键手段 [ 1 - 2 ] 。随着人工智能技术的发展,基于神经网络的医学图像智能分割得到广泛应用 [ 3 - 4 ] ,已经成为人工智能辅助诊疗的重要技术 [ 5 - 6 ] 。
通过人工智能方法进行医学图像分割的过程中,域转移成为一个重要挑战 [ 7 ] 。这主要是由于通过源域训练好的分割模型是针对某种特定类型序列图像的,在其他类型序列图像上分割相同组织时会不匹配,从而导致精度下降。精度误差通常与源域和目标域之间的分布差异成比例。因此,通过源域数据训练的模型,在对目标域数据分割时常常不能达到期望的效果。但研究人员们希望无论数据来自哪个域,都可以使用一个通用模型获得有效的分割结果。解决域转移问题,成为医学图像分析中的迫切需求。
为了应对域转移挑战,研究者提出了域自适应方法 [ 8 ] ,较为常见的为图像级域自适应和特征级域自适应。要使分割网络适应目标域图像,可以通过使用有标签的目标域数据对分割网络进行调整来实现,但这需要对目标域数据进行注释。而图像分割需要进行像素级注释,与其他任务(如图像分类)相比,为分割任务注释数据要困难得多,从而导致分割任务图像通常缺乏人工标签。因此,无监督的域自适应方法受到越来越多的关注。
从图像层面进行的域自适应称为图像级域自适应,经常使用生成对抗网络(generative adversarial network,GAN)作为框架 [ 9 - 11 ] ,尤其是Zhu等 [ 12 ] 提出的CycleGAN,它可以将一个图像转换成另一种图像风格,而不需要成对的训练样本。Wollmann等 [ 13 ] 提出了一种基于CycleGAN的乳腺癌分类方法,首先使用CycleGAN将淋巴结的全切片图像从源域转换到目标域,然后将DenseNet用于乳腺癌分类。Hiasa等 [ 14 ] 基于CycleGAN并结合边缘损失将全髋关节磁共振(magnetic resonance,MR)图像转化为电子计算机断层扫描(computed tomography,CT)图像,并使用U-Net对生成的CT图像进行分割。但他们使用的非端到端的分割方法,不仅推理时间长,而且相对来说训练也更加复杂。此外,Liu等 [ 15 ] 同样使用CycleGAN将不同类型的膝关节MR图像序列进行转化和分割,但他们使用的2维卷积神经网络结构会限制单个切片内的图像特征,并且当图像之间的对比度不一致时,可能会导致分割偏差。
从特征层面进行的域自适应称为特征级域自适应,其主要目的是将源域数据和目标域数据映射到同一个特征空间,促使生成器学习跨域特征。Javanmardi等 [ 16 ] 提出了基于GAN的域自适应方法,并在公开的眼血管数据集和电子显微镜神经元图像分割数据集中进行了实验,将U-Net作为生成器用于图像的分割,使用另一个卷积神经网络作为鉴别器对分割结果进行分类,使用0或者1进行标记。Panfilov等 [ 17 ] 同样基于GAN对膝关节软骨进行了分割,不仅如此,实验中使用了两个鉴别器对不同阶段的特征进行分类,以在高维特征层面进行特征自适应学习,并且在三个数据集上进行了实验,在股骨软骨和胫骨软骨上最好的分割准确度结果提高了3.0%和6.8%。但此工作只对软骨进行了分割,且使用的源域与目标域数据均来自同一公司生产的设备,MR图像间差异较小。
为了更加有效地解决域转移问题,提高分割网络自适应性,本文提出了一种基于GAN的无监督端到端特征级域自适应医学图像分割方法。同时对比分析了本文提出的特征级域自适应方法与三种经典方法:① 直接使用由源域数据训练的分割网络对目标域图像进行分割;② Zhu等 [ 12 ] 于2017年提出的图像级域自适应方法;③ Panfilov等 [ 17 ] 于2019年提出的特征级域自适应方法。进一步的,使用来自不同MR设备且灰度值存在显著差异的膝关节MR图像数据集进行域自适应实验,对所提域自适应方法进行了有效性验证。
1. 特征级域自适应图像分割方法设计
本文设计了一种无监督端到端特征级域自适应图像分割方法,整体流程如
图1
所示(
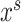 :源域数据,
:源域数据,
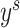 :源域分割标签,
:源域分割标签,
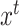 :目标域数据,
:目标域数据,
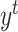 :目标域分割标签)。针对医学图像分割中人工标注工作繁琐、主观性强等问题,我们采用GAN针对目标域数据进行无监督学习训练。我们所提的无监督训练是指在解决域转移问题过程中,只使用目标域数据而未使用目标域标签。GAN中生成器的梯度更新信息来自鉴别器,而不是来自数据样本,同时我们将源域标签作为鉴别器输入,使其更加适合针对目标域数据的无监督训练。本文采用了以残差模块为基本模块的分割网络,增强了对特征的复用能力。通过源域数据进行分割网络预训练,获得适合源域数据的参数作为GAN中生成器即分割网络的参数初值。鉴别器结构为卷积神经网络,其中目标域数据通过重采样与中心裁剪匹配源域数据格式,与源域分割标签的独热编码一起作为鉴别器的输入。再通过GAN对分割网络参数进行学习和调整,获得适合目标域的分割网络参数。随后通过戴斯相似性系数(Dice similarity coefficient,DSC)进行分割精度检验。本文所设计无监督域自适应方法基于Pytorch
[
18
]
实现。
:目标域分割标签)。针对医学图像分割中人工标注工作繁琐、主观性强等问题,我们采用GAN针对目标域数据进行无监督学习训练。我们所提的无监督训练是指在解决域转移问题过程中,只使用目标域数据而未使用目标域标签。GAN中生成器的梯度更新信息来自鉴别器,而不是来自数据样本,同时我们将源域标签作为鉴别器输入,使其更加适合针对目标域数据的无监督训练。本文采用了以残差模块为基本模块的分割网络,增强了对特征的复用能力。通过源域数据进行分割网络预训练,获得适合源域数据的参数作为GAN中生成器即分割网络的参数初值。鉴别器结构为卷积神经网络,其中目标域数据通过重采样与中心裁剪匹配源域数据格式,与源域分割标签的独热编码一起作为鉴别器的输入。再通过GAN对分割网络参数进行学习和调整,获得适合目标域的分割网络参数。随后通过戴斯相似性系数(Dice similarity coefficient,DSC)进行分割精度检验。本文所设计无监督域自适应方法基于Pytorch
[
18
]
实现。
Flow chart of the proposed domain adaptation method
本文提出的域自适应方法流程图
生成器
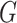 使用之前在我们的工作中提出的分割网络
[
5
]
,具体如
图2
所示。分割网络的基本模块为残差模块,这样设计既增加了网络对特征的复用能力,又起到了降低优化难度的作用。利用并行的扩张卷积模块获取不同的感受野,克服了U-Net模型中单一感受野的局限性,有效提高分割能力的鲁棒性。使用空洞空间金字塔池化模块(atrous spatial pyramid pooling,ASPP),利用具有不同感受野的卷积操作,获取丰富的上下文信息。同时多输出融合的深监督模块能直接利用不同层次的特征,实现多尺度特征信息互补,最终提高了分割区域的连贯性和准确性。
使用之前在我们的工作中提出的分割网络
[
5
]
,具体如
图2
所示。分割网络的基本模块为残差模块,这样设计既增加了网络对特征的复用能力,又起到了降低优化难度的作用。利用并行的扩张卷积模块获取不同的感受野,克服了U-Net模型中单一感受野的局限性,有效提高分割能力的鲁棒性。使用空洞空间金字塔池化模块(atrous spatial pyramid pooling,ASPP),利用具有不同感受野的卷积操作,获取丰富的上下文信息。同时多输出融合的深监督模块能直接利用不同层次的特征,实现多尺度特征信息互补,最终提高了分割区域的连贯性和准确性。
Structure of the generator
生成器结构
鉴别器 D 的结构如 图3 所示,本结构中鉴别器共由5个卷积层组成,初始卷积核数量设置为64,第二层卷积核个数为128,随后逐层加倍,最后一层输出通道被设置为1,再通过双线性插值将最后一层的输出还原至384×384,即原始图像的分辨率大小。所使用的激活函数采用Leaky ReLU。
Structure of the discriminator
鉴别器结构
生成器的具体训练流程如
图4a
所示,源域图像
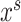 与目标域图像
与目标域图像
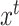 作为生成器输入,生成器输出图像分割结果。损失函数设置如公式(1)所示,由分割损失与对抗损失两部分组成。
作为生成器输入,生成器输出图像分割结果。损失函数设置如公式(1)所示,由分割损失与对抗损失两部分组成。
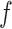 使用
softmax
函数,
使用
softmax
函数,
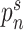 为分割结果的概率值。
为分割结果的概率值。
Training process of the GAN
生成对抗网络训练过程
a. 生成器训练流程; b. 鉴别器训练流程
a. the generator training process; b. the discriminator training process
鉴别器的具体训练流程如 图4b 所示,本文将源域数据分割标签的独热编码作为鉴别器输入之一,选取二分类交叉熵作为其损失函数,鉴别器的输出是与图像分辨率一致的矩阵。二分类交叉熵损失为:
对于源域数据,本文中将其标签设定为矩阵0,对于本地数据其标签为矩阵
1
。将
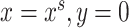 和
和
 分别代入交叉熵损失后相加,可得鉴别器损失函数。且式中
分别代入交叉熵损失后相加,可得鉴别器损失函数。且式中
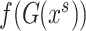 为由生成器得到的源域图像分割标签概率图,我们将此替换为源域标签
为由生成器得到的源域图像分割标签概率图,我们将此替换为源域标签
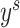 的独热编码,设计了如式(3)所示的鉴别器损失函数,以增加鉴别器精度。
的独热编码,设计了如式(3)所示的鉴别器损失函数,以增加鉴别器精度。
2. 实验数据与实验设计
膝骨关节炎是一种慢性退行性关节疾病,会导致患者关节疼痛和运动障碍。关节软骨的体积和厚度是膝骨关节炎检测和分级的重要指标之一 [ 19 ] 。MR成像长期以来被认为是最强大的膝关节检查工具 [ 20 ] ,临床上通常用于研究膝关节,特别是关节软骨的内部结构变化。对于需要进行膝关节置换术的晚期膝骨关节炎患者,利用分割后的MR图像进行三维重建可以构建患者的膝关节模型,用于个性化假体设计和术前规划 [ 21 ] 。但是由于成像设备差异、同一设备扫描参数差异等原因,域转移问题在膝骨关节炎MR图像的分割中经常出现,并仍未得到有效解决。因此,本文选取了来自不同MR设备且灰度值存在显著差异的膝关节MR图像数据集进行了域自适应方法验证实验。
设计实验,对比分析本文所提特征级域自适应方法和三种经典方法:① 直接使用由源域数据训练的分割网络对目标域图像进行分割;② Zhu等 [ 12 ] 于2017年提出的图像级域自适应方法;③ Panfilov等 [ 17 ] 于2019年提出的特征级域自适应方法。
2.1. 实验数据
本文使用膝关节MR图像公开数据集OAI-ZIB [ 22 ] 作为源域数据,使用由北京大学第三医院运动医学研究所提供并授权使用的膝骨关节炎患者的膝关节MR图像数据集 [ 5 ] 作为目标域数据,进行域自适应方法验证实验,对所提方法的有效性进行验证。
OAI-ZIB数据集来自美国国家卫生研究院,数据集的手工分割由柏林Zuse研究机构进行。手工分割区域包括股骨、股骨软骨、胫骨和胫骨软骨。合作医院提供的本地MR图像数据集,由影像科医师进行了标注,并使用ITK SNAP软件进行分割 [ 23 ] 。具体参数如 表1 所示,两个数据集的MR图像之间存在明显的域转移问题。
表 1. Comparison of the source domain dataset and the target domain dataset with domain shift.
存在域转移问题的源域与目标域数据集对比
公开数据集中的MR图像和合作医院提供的临床数据集MR图像对比如 图5 所示。由于MR图像的具体类型不同,公开数据集图像中骨性区域一般为黑色,软骨区域为白色,而本地数据集则相反,两个数据集中的图像灰度值存在明显差异。
Image comparison of different data sets
不同数据集图像对比
2.2. 实验设备与数据预处理
实验中使用的计算机CPU为E5-2678 v3,内存为128 GB,显卡为2080Ti。
本文使用了OAI-ZIB公开数据集中的1 840张图像以及本地数据集中的1 694张图像,为避免同一病例的MR图像层间相关性对实验造成影响,数据均以每个病例为整体进行划分。实验设计具体为:取OAI-ZIB数据集中的1 840张图像作为源域数据,随机选取本地数据集中2例患者共225张图像作为测试集,1例患者110张图像作为验证集,其余12例患者共1 359张图像作为训练集。由于公开数据集图像与本地数据集图像的像素间距不同,我们对本地数据集图像进行了重采样,采用规定化像素间距方法使本地数据集图像分辨率变为444×444,随后进行中心裁剪使分辨率为384×384,与公开数据集图像分辨率相同。
2.3. 对比实验设计
首先使用源域数据与标签对本文所提出的分割网络进行训练,得到适用于源域数据的分割网络参数。进而使用训练所得参数配置分割网络,进行目标域图像分割,以此作为基线。
其次使用Zhu等 [ 12 ] 提出的CycleGAN图像级域自适应网络,将不同类型的MR图像相互转换。具体为使用通过CycleGAN训练好的生成器,将目标域图像转换为源域图像风格,并输入使用源域数据训练的分割网络。本文遵循之前研究中CycleGAN的实验参数设置,训练生成器和鉴别器。
最后设计与Panfilov等
[
17
]
提出的特征级域自适应方法进行对比。为了控制影响分割结果的因素,对比实验中的参数设定与本文提出的方法相同。批处理大小设置为1,学习率设置为0.001,动量设置为0.9,权重衰减设置为0.000 5,损失函数中
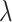 设置为0.1。其中使用公开数据库数据预训练模型,优化器为随机梯度下降(stochastic gradient descent,SGD)
[
24
]
。参数随机初始化后训练鉴别器,并在其中使用了谱标准化
[
25
]
,选用Adam优化器
[
26
]
,学习率设置为0.001。学习率使用Poly更新,次方数0.9,训练次数设定为100 000次。
设置为0.1。其中使用公开数据库数据预训练模型,优化器为随机梯度下降(stochastic gradient descent,SGD)
[
24
]
。参数随机初始化后训练鉴别器,并在其中使用了谱标准化
[
25
]
,选用Adam优化器
[
26
]
,学习率设置为0.001。学习率使用Poly更新,次方数0.9,训练次数设定为100 000次。
3. 模型训练与结果分析
本文将所提特征级域自适应方法与三种经典方法分别进行了训练与分割实验,同时也对CycleGAN在本文所用数据集中图像风格转化的有效性进行了验证。为了直观地展示各种方法在图像分割结果上的差异,随机选取了两幅目标域图像进行不同方法的分割结果定性对比。最后采用戴斯相似性系数对各分割方法在测试集中的分割精度进行了量化评估与对比分析。
3.1. 定性分析
我们在测试集中随机选取两张图像进行对比分析,所提方法与三种已有方法的分割结果如 图6 所示。由于存在域转移问题,直接使用由源域数据训练的分割网络对目标域图像进行分割,无法对股骨和胫骨进行有效分割,分割区域不完整。Zhu等 [ 12 ] 提出的图像级域自适应方法使用CycleGAN将目标域图像转换为源域图像风格,如 图7 所示,再使用由源域数据训练的分割网络进行分割,能有效改善分割结果,但假阳性区域过多。使用Panfilov等 [ 17 ] 提出的特征级域自适应方法后,分割区域存在较多缺失。而本文所提的特征级域自适应方法的分割结果与手工分割结果最为接近。
Qualitative segmentation results comparison of different methods
不同分割方法对比
图 7.
In image-level domain adaptation, the target domain image is converted to the source domain state
图像级域自适应中,目标域图像转化为源域风格
3.2. 定量评估
由于每个病例的MR图像常由多个存在前后关系的二维图像切片组成,故评估分割精度时,本文选择将每个病例的数据作为整体进行评估而不是逐张进行评估,是一种符合数据情况的评估方法。分割精度评估大多采用戴斯相似性系数,其通常用于计算两个集合间的相似度,适合本研究中的相似度量化评估需求,且最近有研究表明戴斯相似性系数在医学图像分割任务中更为合适 [ 27 ] ,故本研究中采用戴斯相似性系数进行定量评估。戴斯相似性系数的计算结果范围为0%~100%,数值越小表示相似度越差,反之则越好。
实验中的待分割部分包括股骨、胫骨、股骨软骨和胫骨软骨。本文采用戴斯相似性系数对每一个分割区域分别进行评估,随后取平均值。戴斯相似性系数的计算过程如公式(4)所示。
其中, Y 代表真值,即目标域的人工分割结果, P 为分割网络预测的结果。
图像分割对比实验结果如 表2 所示,相对于经典域自适应方法,本文提出的无监督特征级域自适应方法在股骨和胫骨区域分割中戴斯相似性系数分别可以达到92.06%和89.34%;同时股骨软骨的分割精度也得到了较大提升,戴斯相似性系数可达到48.03%;另外,本文所提方法可以获得最高的平均戴斯相似性系数64.18%,相对于直接使用由源域数据训练、图像级域自适应方法 [ 12 ] 和特征级域自适应方法 [ 17 ] 分别提升了48.42%、2.52%和6.10%。直接使用源域数据训练分割模型参数对本地临床图像进行分割,戴斯相似性系数最低,甚至无法分割软骨。使用经典图像级域自适应和经典特征级域自适应后,平均戴斯相似性系数有所提高。使用本文所提方法在胫骨软骨分割中戴斯相似性系数低于经典图像级域自适应方法,但股骨、股骨软骨和胫骨的分割中,戴斯相似性系数均高于经典方法。
表 2. The DSC comparison of different domain adaptation methods.
不同域自适应分割方法戴斯相似性系数对比
训练模型 40.61% 0.00% 22.43% 0.00% 15.76% 图像级域自适应
(Zhu等 [ 12 ] ) 88.82% 40.9% 80.86% 36.07% 61.66% 特征级域自适应
(Panfilov等 [ 17 ] ) 90.42% 36.34% 78.89% 26.68% 58.08% 本文所提特征级
域自适应 92.06% 48.03% 89.34% 27.27% 64.18%
经典图像级域自适应对胫骨软骨分割精度高于特征级域自适应方法,但不论是经典方法还是本文所提方法,对胫骨软骨区域进行分割的戴斯相似性系数普遍偏低。进行图像对比后发现,如 图8a 所示,目标域数据集中胫骨软骨部分的对比度较低,胫骨软骨与胫骨骨性区域界限不明显。通过调整窗位、窗宽增加对比度后可以使其界限较为清晰,但这种做法会造成图像中胫骨部分与旁边的肌肉组织部分相似,如 图8b 所示,影响胫骨骨骼部分的分割,故本文在实验中未对目标域数据进行对比度处理。同时本地数据集均来自膝骨关节炎患者,胫骨软骨区域均存在一定的病理问题,且胫骨软骨区域在每张MR图像中占比较小,约为1.7%,特征级域自适应方法在特征学习过程中会受到模糊边界与软骨区域病理问题的影响,从而损失一定的分割精度。
MR images with different contrasts in our dataset
不同对比度的本地数据集图像
a. 原始图像;b. 调整对比度后的图像
a. original image; b. contrast adjusted image
相对于对比方法中的经典域自适应方法,本文所提方法的戴斯相似性系数有一定的提升,骨骼区域由于面积较大几乎在每一张MR图像上都存在,在实际的人工标注中耗时较多,本文所提的域自适应方法可以较好地解决在骨骼区域的域转移问题。
4. 结论
本文针对医学图像智能化分割面临的域转移挑战,提出了一种基于GAN的无监督端到端特征级域自适应图像分割方法。在膝关节MR图像公开数据集和我们合作医院的膝关节MR图像临床数据集上进行了域自适应实验,结果表明所提方法能有效提高分割模型的域自适应能力,显著提高了对胫骨和股骨的分割精度。本文使用膝关节MR图像进行方法验证,此方法还可推广至其他部位的医学图像分割,如髋关节等。未来将开发结合图像级域自适应与特征级域自适应的方法,结合两者优势,同步提高骨性区域与软骨区域的分割精度,从而更好地在临床中为假体设计与术前规划等提供帮助。同时,MR图像是由多个存在前后关系的二维图像切片组成,这些切片包含丰富的结构信息,在以后的工作中,将会基于GAN在无监督训练上的优势,研发基于图像切片间相关性的域自适应图像分割方法,进一步提高分割精度。
重要声明:
利益冲突声明:本文全体作者均声明不存在利益冲突。
作者贡献声明:孙玉波主要负责研究框架设计、方法设计与实现、数据处理与分析、文稿写作以及最后定稿。刘嘉男主要负责方法设计与实现、对比方法代码设计与实现、数据集处理与分析以及文稿写作。孙泽文主要负责数据集整理、打标、分析与解释。韩建达教授与于宁波教授主要负责课题构思与设计、资料的分析和解释、对文稿关键方法与学术内容的修改以及最后定稿。
Funding Statement
国家自然科学基金(U1913208,61873135,61720106012);国家重点研发计划(2018YFB1307803)
References
1. Chowdhary C L, Acharjya D P Segmentation and feature extraction in medical imaging: a systematic review. Procedia Comput Sci. 2020;167:26–36. doi: 10.1016/j.procs.2020.03.179. [ DOI ] [ Google Scholar ] 2. Hesamian M H, Jia W, He X, et al Deep learning techniques for medical image segmentation: achievements and challenges. J Digit Imaging. 2019;32(4):582–596. doi: 10.1007/s10278-019-00227-x. [ DOI ] [ PMC free article ] [ PubMed ] [ Google Scholar ] 3. 吴玉超, 林岚, 王婧璇, 等 基于卷积神经网络的语义分割在医学图像中的应用. 生物医学工程学杂志. 2020;37(3):533–540. doi: 10.7507/1001-5515.201906067. [ DOI ] [ PMC free article ] [ PubMed ] [ Google Scholar ] 4. 王玉丽, 赵子健 基于深度学习的脑图像分割算法研究综述. 生物医学工程学杂志. 2020;37(4):721–729. doi: 10.7507/1001-5515.201912050. [ DOI ] [ PMC free article ] [ PubMed ] [ Google Scholar ] 5. 于宁波, 刘嘉男, 高丽, 等 基于深度学习的膝关节MR影像自动分割方法. 仪器仪表学报. 2020;41(6):140–149. [ Google Scholar ] 6. Aslan M F, Unlersen M F, Sabanci K, et al CNN-based transfer learning–BiLSTM network: A novel approach for COVID-19 infection detection. Appl Soft Comput. 2021;98:106912. doi: 10.1016/j.asoc.2020.106912. [ DOI ] [ PMC free article ] [ PubMed ] [ Google Scholar ] 7. Torralba A, Efros A A Unbiased look at dataset bias// CVPR 2011. Colorado Springs: IEEE. 2011:1521–1528. [ Google Scholar ] 8. Guan H, Liu M Domain adaptation for medical image analysis: a survey. IEEE Trans Biomed Eng. 2021;69(3):1173–1185. doi: 10.1109/TBME.2021.3117407. [ DOI ] [ PMC free article ] [ PubMed ] [ Google Scholar ] 9. Yi X, Walia E, Babyn P Generative adversarial network in medical imaging: A review. Med Image Anal. 2019;58:101552. doi: 10.1016/j.media.2019.101552. [ DOI ] [ PubMed ] [ Google Scholar ] 10. Patgiri R, Biswas A, Roy P Health informatics: A computational perspective in healthcare. Berlin: Springer. 2021:77–96. [ Google Scholar ] 11. Goodfellow I, Pouget-Abadie J, Mirza M, et al Generative adversarial networks. Commun ACM. 2020;63(11):139–144. doi: 10.1145/3422622. [ DOI ] [ Google Scholar ] 12. Zhu J Y, Park T, Isola P, et al Unpaired image-to-image translation using cycle-consistent adversarial networks// Proceedings of the IEEE International Conference on Computer Vision. Venice: IEEE. 2017:2223–2232. [ Google Scholar ] 13. Wollmann T, Eijkman C S, Rohr K Adversarial domain adaptation to improve automatic breast cancer grading in lymph nodes// 2018 IEEE 15th International Symposium on Biomedical Imaging. Washington: IEEE. 2018:582–585. [ Google Scholar ] 14. Hiasa Y, Otake Y, Takao M, et al Cross-modality image synthesis from unpaired data using CycleGAN// Gooya A, Goksel O, Oguz I, et al. International workshop on simulation and synthesis in medical imaging. Cham: Springer. 2018:31–41. [ Google Scholar ] 15. Liu F SUSAN: segment unannotated image structure using adversarial network. Magn Reson Med. 2019;81(5):3330–3345. doi: 10.1002/mrm.27627. [ DOI ] [ PMC free article ] [ PubMed ] [ Google Scholar ] 16. Javanmardi M, Tasdizen T Domain adaptation for biomedical image segmentation using adversarial training// 2018 IEEE 15th International Symposium on Biomedical Imaging. Washington: IEEE. 2018:554–558. [ Google Scholar ] 17. Panfilov E, Tiulpin A, Klein S, et al Improving robustness of deep learning based knee MRI segmentation: Mixup and adversarial domain adaptation// Proceedings of the IEEE/CVF International Conference on Computer Vision Workshops. Seoul: IEEE/CVF. 2019:450–459. [ Google Scholar ] 18. Paszke A, Gross S, Massa F, et al Pytorch: An imperative style, high-performance deep learning library. Adv Neural Inf Process Syst. 2019;32:1–12. [ Google Scholar ] 19. Razmjoo A, Caliva F, Lee J, et al T2 analysis of the entire osteoarthritis initiative dataset. J Orthop Res. 2021;39(1):74–85. doi: 10.1002/jor.24811. [ DOI ] [ PubMed ] [ Google Scholar ] 20. Hayashi D, Roemer F W, Guermazi A Magnetic resonance imaging assessment of knee osteoarthritis: current and developing new concepts and techniques. Clin Exp Rheumatol. 2019;37(Suppl 120):88–95. [ PubMed ] [ Google Scholar ] 21. Gao L, Ding K, Liu J, et al Design and Development of A Knee Surgery Planning System// 2020 Chinese Control And Decision Conference (CCDC) Hefei: IEEE. 2020:2053–2059. [ Google Scholar ] 22. Ambellan F, Tack A, Ehlke M, et al Automated segmentation of knee bone and cartilage combining statistical shape knowledge and convolutional neural networks: Data from the Osteoarthritis Initiative. Med Image Anal. 2019;52:109–118. doi: 10.1016/j.media.2018.11.009. [ DOI ] [ PubMed ] [ Google Scholar ] 23. Yushkevich P A, Gerig G ITK-SNAP: an intractive medical image segmentation tool to meet the need for expert-guided segmentation of complex medical images. IEEE Pulse. 2017;8(4):54–57. doi: 10.1109/MPUL.2017.2701493. [ DOI ] [ PubMed ] [ Google Scholar ] 24. Cherry J M, Adler C, Ball C, et al SGD: Saccharomyces genome database. Nucleic Acids Res. 1998;26(1):73–79. doi: 10.1093/nar/26.1.73. [ DOI ] [ PMC free article ] [ PubMed ] [ Google Scholar ] 25. Miyato T, Kataoka T, Koyama M, et al. Spectral normalization for generative adversarial networks. arXiv preprint arXiv: 2018: 1802.05957. 26. Kingma D P, Ba J. Adam: A method for stochastic optimization. arXiv preprint arXiv: 2014: 1412.6980. 27. Ma J, Chen J, Ng M, et al Loss odyssey in medical image segmentation. Med Image Anal. 2021;71:102035. doi: 10.1016/j.media.2021.102035. [ DOI ] [ PubMed ] [ Google Scholar ]Articles from Sheng Wu Yi Xue Gong Cheng Xue Za Zhi = Journal of Biomedical Engineering are provided here courtesy of West China Hospital of Sichuan University