谁说Python不能绘制环形基因组图?快看这个..

前言
今天是我的可视化课程上线的第210天,目前学员408人,每篇原创公众号都会记录这个人数,用来督促和激励我的原创内容。目前课程的主要方向是 科研、统计、地理相关的学术性图形绘制方法,后续也会增加商务插图、机器学等、数据分析等方面的课程。
参与课程的你将获取到:学员答疑、可视化资源分享、可视化技巧补充、可视化业务代做(学员和甲方对接)、副业交流、提升认知等等。
pyCircos可视化包推荐
Circos 是可视化基因组相似性和特征的最流行软件之一。不过,它的执行过程比较复杂,需要多个原始配置文件才能实现可视化。此外,Circos 是用 Perl 编写的,这限制了它与其他生物分析软件的整合。
另一方面,Python 已被用于各种生物软件包。因此,结合这些软件包,研究人员可以完成大部分所需的分析工作。然而,尽管 Circos 软件已经开发了十多年,Python 却缺少绘制 Circos 图的库。在此,我们提供了一个基于 Python Matplotlib 的环形基因组可视化软件包-pyCircos。用户只需指定注释序列文件(如 GenBank 文件),就能方便快捷地可视化基因组特征和比较基因组分析结果。
- 安装方式
pyCircos可通过如下语句进行快速安装(建议)。
pip install python-circos
如果想安装开发版本,则使用如下语句:
pip install git+https://github.com/ponnhide/pyCircos.git
- 案例教程
- 案例01
#linkplot
#heatmap
values_all = []
arcdata_dict = collections.defaultdict(dict)
with open("sample_data/example_data_links.csv") as f:
f.readline()
for line in f:
line = line.rstrip().split(",")
name1 = line[0]
start1 = int(line[1])-1
end1 = int(line[2])
name2 = line[3]
start2 = int(line[4])-1
end2 = int(line[5])
source = (name1, start1, end1, 615)
destination = (name2, start2, end2, 615)
circle.chord_plot(source, destination, facecolor=circle.garc_dict[name1].facecolor)
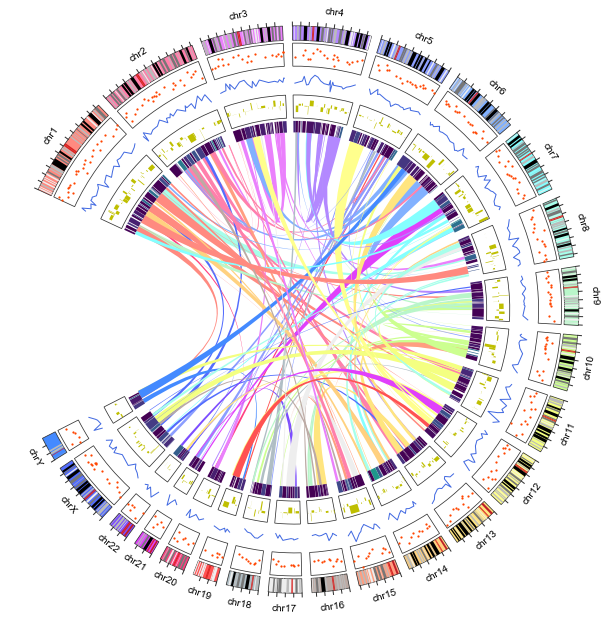
- 案例02
#Plot GCskew
import copy
skews = garc.calc_nnskew(n1="G", n2="C")
positive_skews=copy.deepcopy(skews)
positive_skews[skews<0]=0
negative_skews=copy.deepcopy(skews)
negative_skews[skews>=0]=0
gcircle.fillplot("NC_000913", positive_skews, rlim=(min(skews),max(skews)), base_value=0, raxis_range=(400,700), facecolor="r")
gcircle.fillplot("NC_000913", negative_skews, rlim=(min(skews),max(skews)), base_value=0, raxis_range=(400,700), facecolor="b")
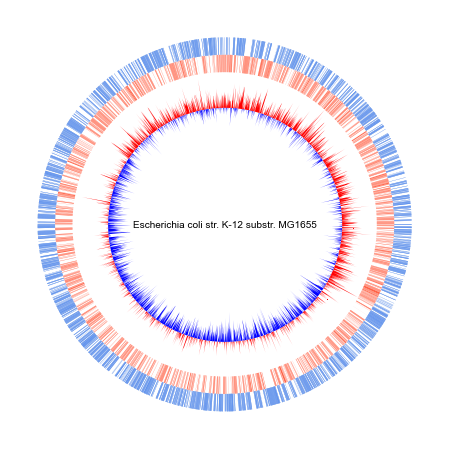
- 案例03
import collections
chord_dict = collections.defaultdict(list)
with open("sample_data/segdup.txt","r") as f:
for line in f:
line = line.rstrip().split("\t")
